Progettazione del progetto: Qualifying Variant Evidence Standard (QV-ES)
Ultimo aggiornamento: 20251123
TLDR: I sistemi sanitari, la ricerca e l’industria non possono scambiarsi risultati genomici perché ogni pipeline produce output differenti. QV-ES risolve questo problema con una specifica di regole condivisa a livello nazionale e uno strato minimo di evidenza che ogni pipeline può generare e che ogni istituzione può verificare.
Perché ne ho bisogno?

- Sono una scienziata o uno scienziato clinico e voglio confermare rapidamente se una variante riportata è affidabile e conforme agli standard di accreditamento.
- Sono una o un data engineer e voglio uno strato di evidenza stabile che si integri facilmente con i miei sistemi di dati.
- Lavoro nell’industria e voglio un formato di output condiviso che protegga i miei metodi e rimanga comunque verificabile.
- Sono una cittadina o un cittadino e voglio risultati chiari dalla scienza e dalla medicina avanzata, con pieno rispetto dei miei dati personali.
Come viene risolto?
Nome del sistema: QV Evidence Framework
Standard di evidenza: QV Evidence Standard (QV-ES)
Il QV Evidence Standard (QV-ES) è uno dei pilastri di un framework più ampio. Il framework comprende tre componenti: la specifica delle regole QV, il registro QV e il QV-ES, che definisce l’evidenza minima verificabile necessaria per l’interpretazione genetica clinica. Gli insiemi QV permettono di separare chiaramente le variabili dell’analisi genetica dal software che le utilizza.
La specifica delle regole fornisce il formato YAML o JSON, il registro conserva gli insiemi di regole QV come oggetti versionati, e QV-ES definisce le regole di evidenza che le pipeline utilizzano per produrre output adatti a database relazionali o a grafo su scala nazionale, come PostgreSQL e RDF.
I fornitori di analisi del genoma generano i risultati delle varianti per l’interpretazione. Per verificarli e fidarsi di tali risultati, il framework applica una logica inversa: misura quanta evidenza verificabile invaliderebbe una interpretazione. Le aziende private e la ricerca pubblica ottengono così una metrica di evidenza condivisa, mantenendo separate le metodologie e la proprietà intellettuale interne.
Documentazione tecnica
Il documento di progettazione open source per questo progetto è stato preparato da Switzerland Omics ed è disponibile all’indirizzo:
https://docs.switzerlandomics.ch/pages/design_qv_evidence_flag.
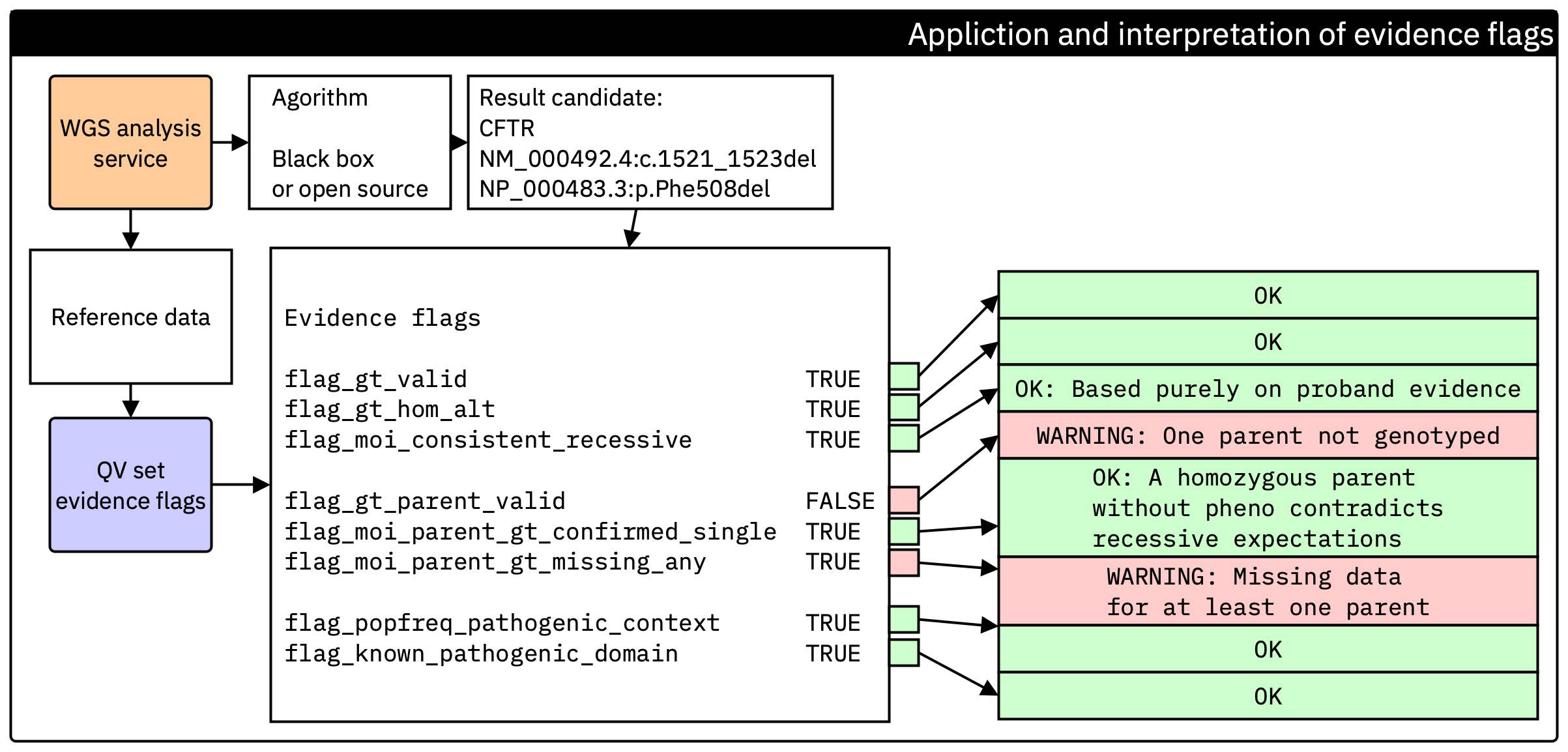 Figura. Generazione dell’insieme di flag di evidenza al termine dell’analisi secondaria. Ogni flag è calcolato direttamente dai dati di riferimento e dai controlli standard, indipendentemente dall’algoritmo di chiamata. Questi flag sono poi trasmessi all’interpretazione terziaria affinché gli utenti finali possano verificare l’evidenza essenziale senza accedere alla pipeline a monte.
Figura. Generazione dell’insieme di flag di evidenza al termine dell’analisi secondaria. Ogni flag è calcolato direttamente dai dati di riferimento e dai controlli standard, indipendentemente dall’algoritmo di chiamata. Questi flag sono poi trasmessi all’interpretazione terziaria affinché gli utenti finali possano verificare l’evidenza essenziale senza accedere alla pipeline a monte.
Il nostro obiettivo
La SGA si concentra sulla pubblicazione del manoscritto ufficiale delle linee guida, attualmente disponibile sulla
nostra pagina delle release:
- Linee guida: Linee guida di consenso della Swiss Genomics Association per l’interpretazione delle varianti genomiche basata sull’evidenza nelle malattie mendeliane.
About (this page) | PDF (EN) | Repository


