Conception du projet: Qualifying Variant Evidence Standard (QV-ES)
Dernière mise à jour: 20251123
TLDR : Les systèmes de santé, la recherche et l’industrie ne peuvent pas échanger de résultats génomiques, car chaque pipeline produit des sorties différentes. QV-ES résout ce problème grâce à une spécification de règles commune à l’échelle nationale et une couche minimale d’évidence que chaque pipeline peut générer et que chaque institution peut vérifier.
Pourquoi en ai-je besoin ?

- Je suis scientifique clinique et je souhaite confirmer rapidement si une variante rapportée est fiable et conforme aux exigences d’accréditation.
- Je suis ingénieur ou ingénieure en données et je souhaite une couche d’évidence stable qui s’intègre proprement dans mes systèmes de données.
- Je travaille dans l’industrie et je souhaite un format de sortie commun qui protège mes méthodes tout en restant vérifiable.
- Je suis citoyenne ou citoyen et je souhaite des résultats clairs issus de la science et de la médecine de pointe, avec un respect total de mes données personnelles.
Comment cela est-il résolu ?
Nom du système : QV Evidence Framework
Standard d’évidence : QV Evidence Standard (QV-ES)
Le QV Evidence Standard (QV-ES) constitue un pilier d’un cadre plus large. Ce cadre comprend trois composants : la spécification des règles QV, le registre QV et le QV-ES, qui définit l’évidence vérifiable minimale nécessaire à l’interprétation en génétique clinique. Les ensembles QV permettent de séparer clairement les variables d’analyse génétique du logiciel qui les utilise.
La spécification de règles fournit le format YAML ou JSON, le registre stocke les ensembles de règles QV sous forme d’objets versionnés, et le QV-ES définit les règles d’évidence qui permettent aux pipelines de produire des sorties adaptées aux bases de données relationnelles ou graphiques à grande échelle, comme PostgreSQL et RDF.
Les fournisseurs d’analyse du génome génèrent les résultats de variantes pour l’interprétation. Pour vérifier et fiabiliser ces résultats, le cadre applique une logique inverse : il mesure la quantité d’évidence vérifiable qui invaliderait une interprétation. L’industrie privée et la recherche publique bénéficient ainsi d’une métrique d’évidence commune tout en gardant leurs méthodes internes et leur propriété intellectuelle séparées.
Documentation technique
Le document de conception open source pour ce projet a été préparé par Switzerland Omics et est disponible à l’adresse :
https://docs.switzerlandomics.ch/pages/design_qv_evidence_flag.
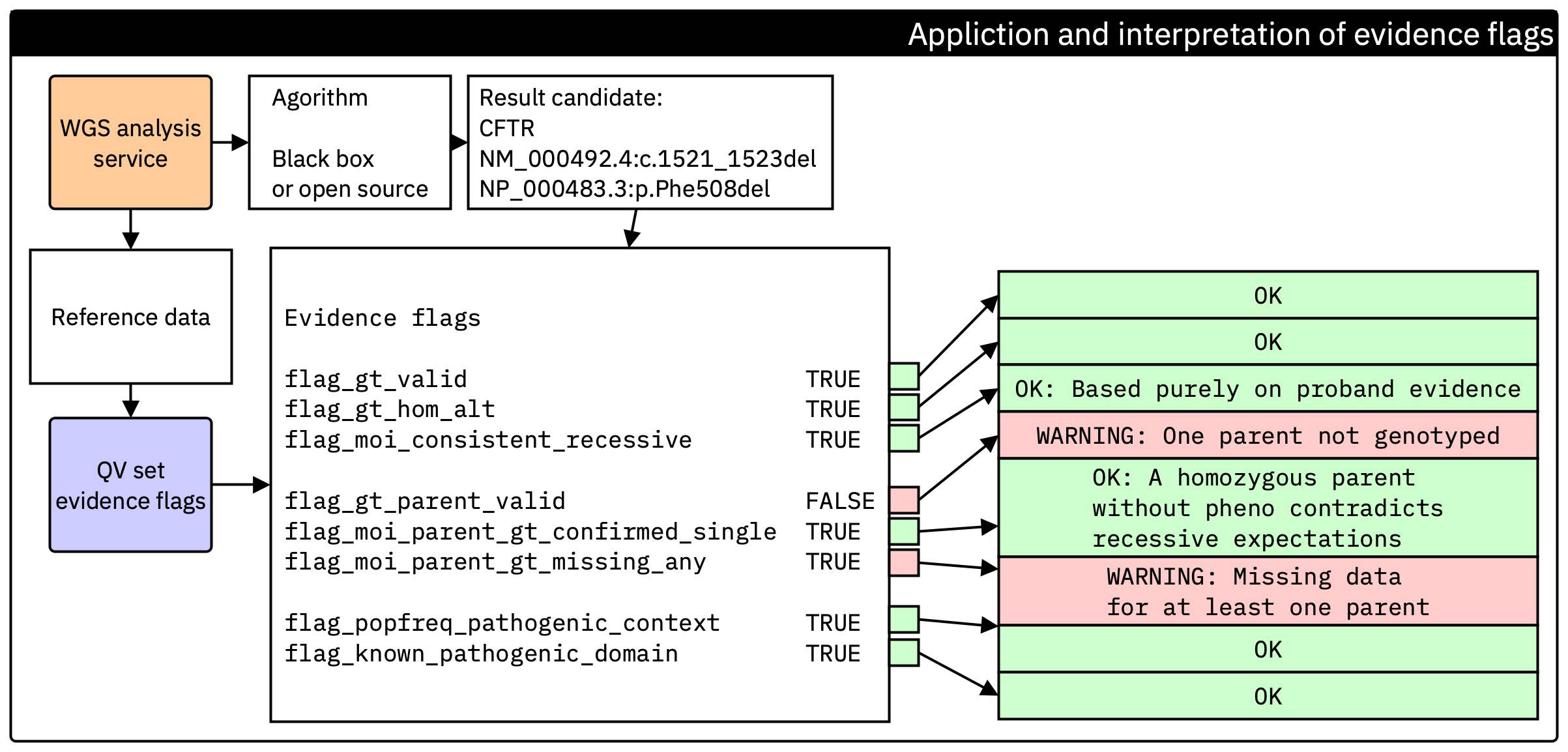 Figure. Génération de l’ensemble des indicateurs d’évidence à la fin de l’analyse secondaire. Chaque indicateur est calculé directement à partir des données de référence et des contrôles standard, indépendamment de l’algorithme d’appel. Ces indicateurs sont ensuite transmis à l’interprétation tertiaire afin que les utilisateurs finaux puissent vérifier l’évidence essentielle sans accéder à la pipeline en amont.
Figure. Génération de l’ensemble des indicateurs d’évidence à la fin de l’analyse secondaire. Chaque indicateur est calculé directement à partir des données de référence et des contrôles standard, indépendamment de l’algorithme d’appel. Ces indicateurs sont ensuite transmis à l’interprétation tertiaire afin que les utilisateurs finaux puissent vérifier l’évidence essentielle sans accéder à la pipeline en amont.
Notre objectif
La SGA se concentre sur la publication du manuscrit officiel des lignes directrices, actuellement disponible sur
notre page des versions :
- Lignes directrices : Lignes directrices consensuelles de la Swiss Genomics Association pour l’interprétation génomique fondée sur l’évidence dans les maladies mendéliennes.
About (this page) | PDF (EN) | Repository


