Projektdesign: Qualifying Variant Evidence Standard (QV-ES)
Letztes Update: 20251123
TLDR: Gesundheitssysteme, Forschung und Industrie können genomische Resultate nicht austauschen, weil jede Pipeline etwas anderes ausgibt. QV-ES löst dies mit einer gemeinsamen Regelspezifikation im nationalen Massstab und einer minimalen Evidenzschicht, die jede Pipeline erzeugen und jede Institution prüfen kann.
Warum brauche ich das?

- Ich bin klinische Wissenschaftlerin oder klinischer Wissenschaftler und möchte schnell bestätigen, ob eine gemeldete Variante zuverlässig und für Akkreditierungsstandards geeignet ist.
- Ich bin Data Engineer und möchte eine stabile Evidenzschicht, die sich sauber in meine Datensysteme integrieren lässt.
- Ich arbeite in der Industrie und möchte ein gemeinsames Ausgabeformat, das meine Methoden schützt und dennoch überprüfbar bleibt.
- Ich bin Bürgerin oder Bürger und möchte klare Resultate aus modernster Wissenschaft und Medizin, wobei meine personenbezogenen Daten respektiert werden.
Wie wird das gelöst?
Systemname: QV Evidence Framework
Evidenzstandard: QV Evidence Standard (QV-ES)
Der QV Evidence Standard (QV-ES) ist eine Säule eines grösseren Frameworks. Das Framework umfasst drei Komponenten: die QV-Regelspezifikation, das QV-Register und den QV-ES, der die minimale überprüfbare Evidenz für die Interpretation in der klinischen Genetik definiert. QV-Sets ermöglichen eine klare Trennung zwischen Analysevariablen und der Software, die sie nutzt.
Die Regelspezifikation definiert das YAML- oder JSON-Format, das Register speichert beide QV-Regelsätze als versionierte Objekte, und QV-ES liefert die Evidenzregeln, die Pipelines verwenden, um Ausgaben zu erzeugen, die für relationale oder Graphdatenbanken im nationalen Massstab geeignet sind, etwa PostgreSQL und RDF.
Genomanalyseanbieter liefern die Variantenresultate zur Interpretation. Um diese Resultate zu prüfen und ihnen zu vertrauen, verwendet das Framework die umgekehrte Logik: Es misst, wie viel überprüfbare Evidenz eine Interpretation ungültig macht. Private Unternehmen und öffentliche Forschung erhalten eine gemeinsame Evidenzmetrik, während interne Methoden und IP getrennt bleiben.
Technische Dokumentation
Das Open-Source-Designdokument für dieses Projekt wurde von Switzerland Omics erstellt und ist direkt unter
https://docs.switzerlandomics.ch/pages/design_qv_evidence_flag verfügbar.
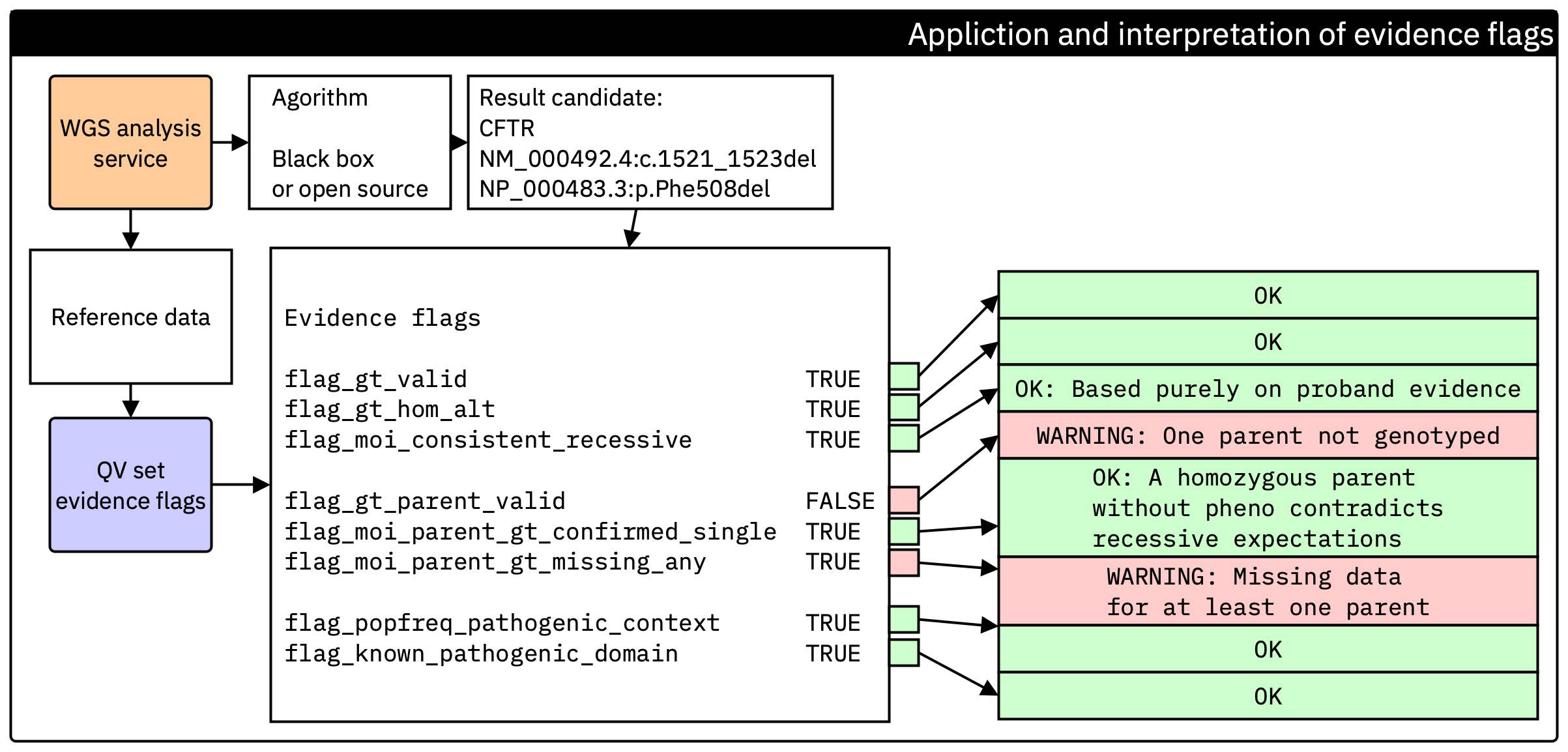 Abbildung. Erzeugung des Evidenz-Flag-Sets am Ende der Sekundäranalyse. Jedes Flag wird direkt aus Referenzdaten und Standardprüfungen berechnet, unabhängig vom Calling-Algorithmus. Diese Flags werden in die tertiäre Interpretation übernommen, sodass Endnutzerinnen und Endnutzer die wesentliche Evidenz ohne Zugriff auf die vorgelagerte Pipeline prüfen können.
Abbildung. Erzeugung des Evidenz-Flag-Sets am Ende der Sekundäranalyse. Jedes Flag wird direkt aus Referenzdaten und Standardprüfungen berechnet, unabhängig vom Calling-Algorithmus. Diese Flags werden in die tertiäre Interpretation übernommen, sodass Endnutzerinnen und Endnutzer die wesentliche Evidenz ohne Zugriff auf die vorgelagerte Pipeline prüfen können.
Unser Ziel
Die SGA konzentriert sich auf die Veröffentlichung des offiziellen Leitfadenmanuskripts, das derzeit auf
unserer Release-Seite verfügbar ist:
- Leitfaden: Swiss Genomics Association Konsensleitfaden für evidenzbasierte genomische Varianteninterpretation bei Mendelschen Erkrankungen.
About (this page) | PDF (EN) | Repository


